ارتباط مستقیم با کارشناس فروش
کشف Microrna , و تاثیر آن در ژن | جایزه نوبل 2024 زیست شناسی

جایزه نوبل زیست شناسی 2024
کشف ریز RNA و نقش آن در تنظیم پس از رونویسی ژنها
مقدمه
miRNA ها دسته ای از RNA های غیر کد کننده هستند که دارای نقش مهمی در تنظیم بیان ژن می باشند. اینRNA های غیر کد کننده کوچک به طور متوسط 22 نوکلئوتید طول دارند. اکثر miRNA ها از توالی های DNA به miRNA های اولیه (pre-miRNA) رونویسی می شوند و به ریز RNA های پیش ساز (pre-miRNA) و miRNA های بالغ پردازش می شوند. در اکثر موارد، miRNA ها با UTR 3 mRNA هدف برای سرکوب بیان تعامل دارند. با این حال، تعامل miRNA ها با سایر مناطق، از جمله UTR 5’، توالی کد کننده و پروموترهای ژن نیز گزارش شده است. علاوه بر این، miRNA ها نشان داده اند که بیان ژن را تحت شرایط خاصی فعال می کنند. مطالعات اخیر نشان دادهاند که ریز RNA ها بین بخشهای مختلف درون سلولی جابهجا میشوند تا سرعت ترجمه و حتی رونویسی را کنترل کنند.
تاریخچه:
کشف اولین microRNA (miRNA)، lin-4، در سال 1993 توسط گروه های Ambros و Ruvkun در Caenorhabditis elegans حوزه زیست شناسی مولکولی را متحول کرد.
در دهه 1980، آمبروس(Ambros) و روکون (Ruvkun) هر دو به عنوان همکاران فوق دکترا در آزمایشگاه رابرت هورویتز کار می کردند. آنها یک کرم گرد به نام Caenorhabditis elegans که به عنوان یک ارگانیسم مدل برای نشان دادن چگونگی تبدیل سلول ها و بافت ها به حیوان استفاده می شود را مرد مطالعه قرار دادند. به طور خاص، این زوج تحقیقاتی را روی کرمهایی انجام دادند که جهشهای خاصی را در DNA خود داشتند که منجر به رشد غیرعادی آنها می شد. به نظر می رسید ژنی به نام lin-4 به نوعی مانع از بیان ژن دیگری به نام lin-14 می شود. این دو دانشمند پس از ترک آزمایشگاه به تحقیقات خود ادامه دادند و متوجه شدند که lin-4 یک RNA کدکننده پروتئین نیست، بلکه در واقع یک RNA کوچک غیر کدکننده است . آنها همچنین دریافتند که lin-14 در مراحل پس از رونویسی از طریق ناحیه 3’UTR کاهش می یابد و اینکه lin-4 توالی مکمل با ناحیه 3’UTRدر lin-14 دارد. بنابراین، آنها عنوان کردند که lin-4، lin-14 را در سطح پس از رونویسی تنظیم می کند. بنابراین lin-4 می تواند از ساخت پروتئین lin-14 جلوگیری کند و روش جدیدی برای روشن و خاموش کردن ژن ها در داخل سلول ها ارائه می دهد. بنظر عجیب و غریب بود تا تا 7 سال بعد، زمانی که روکون یک ریز RNA دیگر به نام let-7 را کشف کرد که برخلاف ریز lin-4 RNA ، به طور گسترده در موجودات در سراسر قلمرو حیوانات یافت شد.
بنابراین ، پس از سالها جایزه نوبل زیست شناسی 2024 در فیزیولوژی یا پزشکی به دلیل کشف microRNA (ریز RNA) و نقش آنها در تنظیم بیان ژن پس از رونویسی به این دو دانشمند یعنی Victor Ambros و Gary Ruvkun اهدا شد.
مکانیسم های تنظیم ژن با واسطه ریز miRNA یا RNA :
ترجمه Mrna یک فرآیند سلولی دقیق در طول رشد و نمو می باشد بنابراین حفظ فرآیندهای فیزیولوژیکی در سلول ضروری می باشد.
کنترل ترجمه نقش اصلی را در تنظیم بیان ژن ایفا می کند که ریز miRNA یا RNA در این تنظیم نقش دارد. اکثر مطالعات تا به امروز نشان داده اند که miRNA ها به یک توالی خاص در ‘ UTR 3 مربوط به mRNA های هدف خود متصل می شوند .
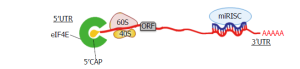
اتصال miRNA به این توالی می تواند منجر به مهار ترجمه یا تخریب Mrna بشود.

سایتهای اتصال miRNA نیز در سایر مناطق mRNA از جمله 5’UTR و توالی کدکننده و همچنین در نواحی پروموتر شناسایی شدهاند.
مسیرهای پردازش ریز RNA یا miRNA :
بیوژنز miRNA در دو مرحله اصلی در هسته و سیتوپلاسم انجام می شود. ژن های miRNA توسط RNA پلیمراز Ⅱ رونویسی شده و از طریق هر دو روش بیوژنز canonical و non-canonical پردازش می شوند. در طول بیوژنز canonical ، miRNA های اولیه (pri-miRNA ها) ابتدا ساخته می شوند .سپس توسطDrosha Ⅲ ribonucleasee و DiGeorge Syndrome Critical Region 8 (DGCR8) به درون هسته پردازش می شوند و یک محصول پیش ساز miRNA ((pre-miRNA تولید می کنند. pre-miRNA به سیتوپلاسم صادر می شود و miRNA بالغ پردازش می شود . در نهایت به همراه پروتئین های خانواده Argonaute (AGO) ، کمپلکس القایی خاموش کننده miRNA miRNA-induced silencing complex (miRISC) را شکل می دهند.
در بیوژنز noncanonical ، pre-miRNA ها توسط سیستم splicing ایجاد می شوند. در این مسیر small hairpin RNA (shRNA) توسط کمپلکس microprocessor برش میخورند و سپس به سیتوپلاسم فرستاده می شوند. در این مسیر پردازش بیشتر از طریق مسیر وابسته به AGO2، اما مستقل از Dicer صورت می گیرد.
خاموش کردن ژن با واسطه MicroRNA از طریق miRISC
برای یک کمپلکس عملکردی miRISC حداقل به حضور پروتئین های miRNA و AGO نیاز است. در واقع مهمترین و بهترین اجزای miRISC پروتئین های خانواده Argonaute هستند.این خانواده در پستانداران دارای چهار نوع پروتئین های آرگونات در پستانداران دارای چهار نوع ,AGO1 AGO2 , AGO3 و AGO4 هستند. پروتئین های AGO میتوانند بدون توجه به توالی نوکلئوتیدی به رشته راهنما متصل شوند. با این حال برخی از AGO ها ترجیح می دهند به توالی خاصی در انتهای 5′ متصل شوند.در اتصال به توالی خاص، کمپلکس خاموش کننده (miRISC) به یک توالی مکمل بر روی mRNA هدف به نام(MREs) miRNA response element متصل می شوند. در سلول های حیوانی، اکثر برهمکنش های miRNA:MRE به طور کامل مکمل نیستند و بیشترMRE ها دارای عدم تطابق مرکزی با miRNA راهنمای خود هستند که از فعالیت اندونوکلئازی AGO2 جلوگیری می کند. در بسیاری از موارد، یک تعامل عملکردی miRNA:MRE از طریق ناحیه 5′ (نوکلئوتیدهای 2-8) صورت می پذیرد.
AGO فاکتورهای دیگری مثل Dicer,TRBP,PACT و TNRC6A-C(GW182) را با کمپلکس همراه می کند. تشکیل یک کمپلکس miRISC خاموش کننده با بکارگیری خانواده پروتئین های ( glycine-tryptophan –rich 182KD ) GW182 توسط miRISC شروع می شود. GW182 داربست مورد نیاز برای شکل گیری miRISC را فراهم میکند . GW182 با AGO ، PAN3، CONT1 ، CONT9 ، زیرواحدهای PAN2/PAN3 و کمپلکس CCR4-NOT میانکنش می دهد.
Poly(A)-binding proteins (PABPs) شامل یک خانواده از پروتئین های متصل شونده به rna (RBPs) که بسیار حفظ شده هستند می باشد که در تنظیم انتقال هسته ای mRNA ،شروع ترجمه ، پایان ترجمه و تنظیم طول دم polyA و تخریب mRNA نقش دارند. حذف PABP یا مهار آن منجر به ناپایداری mRNA می شوند. تعامل مستقیم PABP متصل به پلی (A) با eIF4G منجر به افزایش ترجمه وابسته به eIF4F می شود. کمپلکس( PAN2/PAN3 (PAN2/3)، ریبونوکلئاز مخصوص پلی A ((PAN اولین آنزیم دآدنیلاسیون می باشد.جهشی که منجر به تغییرات در طول دم mRNA poly(A) می شود. تعامل با پروتئین سیتوپلاسمی PABPC منجر به دآد نیلاسیون mRNA پلی(A) به صورت توده ای می شود ، در حالی که برهمکنش با GW182 منجر به دآدنیلاسیون اختصاصی mRNA می شود.
کمپلکس CCR4-NOT یک کاتابولیت مهارکننده کربن چند زیر واحدی 4 (CCR4) – فاقدTATA- در مهار ترجمه القا شده توسط miRNA نقش ایفا می کند. به طور کلی تصور می شود که CCR4-NOT از دو ماژول اصلی تشکیل شده است: (الف) ماژول کاتالیزوری شامل پروتئین های دآدنیلاز CNOT6، CNOT6L، CNOT7 و CNOT8 و (ب) ماژول NOT، حداقل شامل CNOT1، CNOT2 و CNOT3 .
زیر واحدهای غیر کاتالیزوری به طور قابل ملاحظه ای فعالیت دآدنیلاز و اختصاصیت زیرواحدهای کاتالیتیک را با فراهم کردن جایگاه اتصال برای سایر فاکتورهایی که منجر به گردآوری CCR4 به Mrna هدف می شوند را افزایش می دهند. CNOT1 ،زیرواحد داربست این مجموعه، محل اتصال زیرواحدهای دیگر را فراهم می کند.
تشکیل یک کمپلکس miRISC خاموش کننده با بکارگیری خانواده پروتئین های ( glycine-tryptophan –rich 182KD ) GW182 توسط miRISC شروع می شود. GW182 داربست مورد نیاز برای بهکارگیری پروتئینهای مؤثر دیگر را فراهم میکند ، مانند کمپلکسهای پلی (A) – دآدنیلاز، PAN2-PAN3 و CCR4-NOT، به دنبال تعامل miRNA: mRNA . پلی (A) – ددنیلاسیون Mrna هدف توسط PAN2/3 آغاز شده و توسط کمپلکس CCR4-NOT تکمیل می شود. برهمکنش بین تکرارهای تریپتوفان (W) GW182 و پلی (A) پروتئین C (PABPC) باعث ددنیلاسیون کارآمد می شود. به دنبال آن، decapping توسط decapping پروتئین 2 (DCP2) و پروتئینهای مرتبط تسهیل میشود و در نهایت مهار یا تجزیه Mrna هدف صورت می گیرد.
تنظیم ژن رونویسی و پس از رونویسی با واسطه MicroRNA در هسته
به واسطه Importin-8 یا Exportin-1، AGO2 انسانی بین هسته و سیتوپلاسم از طریق تعامل خود با TNRC6A (پروتئین خانواده (GW182 که حاوی سیگنال مکان یابی و خروج از هسته است، جابجا می شود .یافته نشان داده است که miRISC قرار گرفته در هسته هم نرخ رونویسی و هم سطوح پس از رونویسی mRNAرا تنظیم میکند و با یوکروماتین در جایگاههای ژنی با رونویسی فعال مرتبط است . اگرچه، درک ما از زمان و چگونگی عملکرد miRNA ها در هسته هنوز محدود است.
گزارش شده است که miRISC با وزن مولکولی کم می تواند با mRNA های درون هسته تعامل داشته باشد و باعث تخریب mRNA هسته ای شود، اگرچه مکانیسم این امر نامشخص است. عملکرد miRNA در ژن های رونویسی فعال ممکن است نشان دهد که miRISC با mRNA هدف به صورت همزمان/پس از رونویسی تعامل داشته باشد. دخالت AGO و Drosha در mRNA splicing ، از تعاملات miRISC:mRNA درزمان رونویسی پشتیبانی میکند همچنینmiRISC ممکن است رونویسی را مستقیماً تنظیم کند. یک مطالعه نشان داد که AGO2 در هسته فیبروبلاستها متمرکز شده و با miRISC و رتینوبلاستوما (Rb) برای سرکوب رونویسی ژنهای تقویتکننده تکثیر که توسط Rb/E2F تنظیم میشوند، تعامل دارد. اشاره شد که let-7f به MREهای یافت شده در پروموترهای دو ژن هدف E2F، CDCA8 و CDC2، به روشی وابسته به AGO2 متصل است. همچنین، زیرمجموعهای از ژنهای متصل به پروموتر AGO به دنبال پیری افزایش بیان داشته و مشخص شد که AGO2 با یوکروماتین همزمان رسوب ایمنی میکند .
مطالعات نشان داده اند که تنظیم ژن با واسطه miRNA پویا است .در سال های اخیر درک جامع تری از دینامیک miRNA در تنظیم ژن با واسطه miRNA حاصل شده است. عواملی که ممکن است در استحکام تنظیم ژن با واسطه miRNA نقش داشته باشند عبارتند از بخش بندی و جابجایی عملکردی miRISC در داخل سلول ها. در دسترس بودن و فراوانی miRNA ها و mRNA های هدف آنها نیز عواملی هستند که در تعیین اینکه کدام ژن ها تنظیم شود دخیل هستند. اگرچه همیشه اینطور نیست، اما سرکوب miRNA اهداف mRNA در همه جا بین انواع سلول وجود ندارد. Alternative splicing و پلیآدنیلاسیون متغیری که بر 3′ UTRها تأثیر میگذارد، و پروتئینهای اتصال RNA اختصاصی سلولی که بر ساختارهای ثانویه mRNA هدف تأثیر میگذارند، مجموعه موجود MREها را تغییر میدهند. این امر باعث می شود که زیرمجموعه های mRNA ها نسبت به تنظیم ژن با واسطه miRNA به شیوه ای خاص از نوع سلول/اختصاصیت حساس یا غیر حساس باشند.



