ارتباط مستقیم با کارشناس فروش
توالی یابی DNA چیست؟ | روش های توالی یابی | توالی یابی سنگر

توالی یابی DNA ( تعیین سکانس )
توالی یابی DNA یا (DNA Sequencing) فرآیندی است که در آن تعیین ترتیب نوکلئوتیدهای موجود در یک مولکول DNA انجام میگیرد. DNA موجودات زنده متشکل از یک توالی ویژه از نوکلئوتیدها است. تعیین توالی DNA این امکان را به دانشمندان میدهد که با مقایسه DNA بین اندام های مختلف ، ارتباطات بین اندامها و روابط فیلوژنتیکی آنها را بررسی کنند.
گزیده ای از مفاهیم توالی یابی DNA
بطور کلی توالی یابی DNA شناسایی و ترتیب قرارگیری چهار باز نوکلئوتیدی آدنین، گوانین، سیتوزین و تیمین در مولکول است. الزام توالی یابی DNA برای اولین بار توسط نظریه فرانسیس کریک مطرح شد که برپایه این نظریه مبرهن شد که توالی نوکلئوتیدها در یک مولکول DNA مستقیماً بر توالی اسیدهای آمینه پروتئینها اثرگذار است. در آن زمان دانشمندان معتقد بودند که توالی یابی کامل ژنوم، منتهی به جهش بزرگی در فهم بیوشیمیایی سلولها و ارگانیسمها میشود.
در تعیین توالی DNA مدرن، از روشهای پرتوان استفاده میشود که که این امکان را فراهم کردهاند که توالیهای DNA در مدت زمان بسیار کوتاهی مورد شناسایی قرار بگیرند. این فناوری برای بسیاری از شرکتها این امکان را به وجود آورده است تا بتوانند روشهایی برای آزمایشهای خانگی DNA را به مشتریان خود پیشنهاد دهند. بسیاری از نتایج حاصل از این آزمایشها، اختصاصا” ارتباطی است که بین یک واریانت ژنتیکی و یک بیماری خاص وجود دارد. این فناوری همچنین به دانشمندان اجازه داده است که DNA بسیاری از ارگانیسمها را مورد بررسی قرار دهند تا روابط تکوینی و فیلوژنتیکی بین آنها را بهتر مطالعه کنند.

نمونهای از توالی یابی DNA
تعیین توالی DNA در گذشته و در سالهای اول اختراع این فناوری به زمان بسیاری نیاز داشت و گاهی تا چندین سال برای دست یافتن به نتیجه زمان نیاز داشت، اما در حال حاضر با توسعه تکنولوژی، تعیین توالی DNA را میتوان طی چند ساعت انجام داد. جالب است بدانید که اولین پروژه تعیین توالی کامل DNA انسانی حدود سه میلیارد دلار هزینه در بر داشت، در حالی که اکنون، شرکتهای معتبر در این زمینه ، کل ژنوم انسان را با قیمت کمتر از هزار دلار تعیین توالی میکنند و با پیشرفتهترین آزمایشها، نوکلئوتیدهای موجود در ژنوم انسان را مورد تجزیه و تحلیل قرار میدهند. شرکتهای فعال در حوزه ی بیوتکنولوژی و ژنتیکی معتبر در دنیا در کنار توالی یابی DNA ، خدماتی مانند آزمایشهای پلی مورفیسم تک نوکلئوتیدی برای شناسایی و مطالعه ی تغییرات و جهشهای ژنتیکی در ژنوم انسان را نیز عرضه میکنند.
این آزمایشها بر نوکلئوتیدهای اختصاصی در ژنهایی هستند که قادر به نشان دادن واریانت ژنتیکی منحصری هستند. به عبارت دیگر پلی مورفیسم تک نوکلئوتیدی، از آنجایی که در تحقیقات بالینی هم مورد بررسی و مطالعه قرار گرفت، با شرایط خاصی مرتبط هستند و میتوانند در پیشبینی چگونگی تأثیر ژنهای بدن بر زندگی انسان اثرگذار باشند. البته باید توجه داشته باشید که بعضی از توالیهایSNP با یک سری از بیماری های خاص مرتبط هستند، در حالی که بعضی دیگر منوط به متابولیسم بدن و چگونگی پردازش مواد مغذی در آن است. در مورد پلی مورفیسم تک نوکلئوتیدی هزاران رابطه مختلف یافت شده و میتوان تاثیر ژنوم انسان بر زندگی فردی با کمک تعیین توالی DNA استفاده کرد.
روشهای توالی یابی DNA
توالی یابی DNA به دو طریق اصلی در سراسر جهان اعمال میشود. روش قدیمی آن به عنوان روش خاتمه زنجیرهای یا روش سنگر (Sanger Method) معروف است. روشهای جدیدتر که میتوانند تعداد زیادی از مولکولهای DNA را با سرعت بیشتری پردازش کنند، در مجموع به عنوان روش توالی یابی با بازدهی بالا یا روشهای توالی یابی نسل جدید (Next Generation Sequencing) معروف هستند.
توالی یابی به روش سنگر
روش سنگر بر مبنای یک آغازگر پایدار است که به مولکول DNA دناتوره شده (تک رشتهای) متصل میشود و سنتز یک مولکول پلی نوکلئوتیدی تک رشتهای را در حضور یک آنزیم DNA پلیمراز، (با استفاده از DNA دناتوره شده به عنوان رشته الگو) شروع میکند. در بیشتر مواقع ، آنزیم با اضافه کردن نوکلئوتیدها به رشته آغازگر، واکنش را کاتالیز میکند. بنابراین پیوند کووالانسی بین اتم کربن ’3 مولکول قند دئوکسی ریبوز در یک نوکلئوتید و اتم کربن ’5 نوکلئوتید بعدی تشکیل میشود.
در یک مخلوط واکنش توالی یابی، ممکن است یک بخش کوچکی از نوکلئوتیدهای تغییر یافته که به دلیل فقدان گروه واکنشگر هیدروکسیل، نمیتوانند پیوند کووالانسی تشکیل دهند، واکنش تکثیر را متوقف کنند و دی دئوکسی ریبونوکلئوتیدها را ایجاد نکنند. در اصل این نوکلئوتیدها فاقد اتم اکسیژن ’2 یا ’3 را در مقایسه با سایر ریبونوکلئوتیدها هستند. با این کار واکنش پلیمریزاسیون DNA به موقع پایان میگیرد. در پایان چند دور از چنین پلیمریزاسیونهایی، مخلوطی از مولکولها با طولهای مختلف به وجود می آورد.
درمرحله نخست تلاش در روش سنگر، ابتدا مولکول DNA با استفاده از یک آغازگر برچسب دار پشتیبانی شده و سپس به چهار لوله آزمایش تقسیم میشود که هر کدام از این لوله ها یک نوع ddNTP دارند، یعنی هر مخلوط واکنش فقط یک نوع نوکلئوتید تغییر یافته دارد که میتواند منجر خاتمه زنجیره شود. پس از اتمام چهار واکنش، مخلوط مولکولهای DNA ایجاد شده توسط روش خاتمه زنجیره یا سنگر، تحت الکتروفورز با ژل پلی آکریل آمید قرار گرفته و با توجه به طول هر قطعه از یکدیگر جدا میشوند.
شکل زیر ، یک واکنش توالی با ddATP الکتروفورز میشود. هر خط یک مولکول DNA از یک طول خاص را نشان میدهد که در نتیجه یک واکنش پلیمریزاسیون است که با اضافه کردن یک نوکلئوتید ddATP خاتمه یافته است. ستونهای دوم، سوم و چهارم شامل ddCTP ، ddTTP، ddGTP بودند.
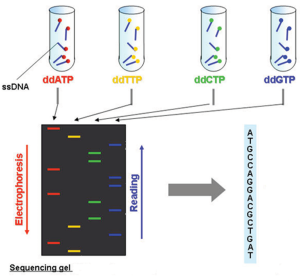
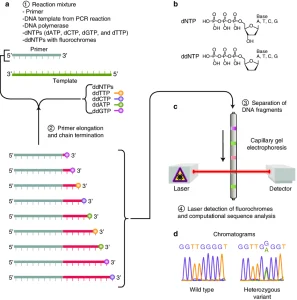
با گذشت زمان، تغییرات این روش با وجود یک برچسب فلورسنت متفاوت در هر ddNTP بود. در واقع برای آغازگر دیگر منبع برچسب رادیواکتیو یا فلورسنت استفاده نشد. بنا براین متد سنگر همان روش توالی یابی رنگ پایان دهنده میباشد، که از چهار رنگ با طیف انتشاری بدون همپوشانی برای هر یک از ddNTP استفاده میکند.
همانطور که در تصویر مشاهده میکنید رنگ خاتمه دهنده از یک طرح کلی توالی یابی را مشاهده میکنید، یک مخلوط واکنش منفرد با تمام فاکتورهای مورد نیاز برای گسترده سازی مولکول DNA را دارد. مخلوط واکنش همچنین حاوی غلظتهای کمی از چهار ddNTPs با برچسب فلورسنت متفاوتی هستند. پس از اتمام واکنش توالی یابی، محصول را بر روی ژل کپیلاری الکتروفورز میبرند و با آنالیز طیف انتشار از هر باند DNA روی ژل نتایج این فرایند بدست میآید. سپس با استفاده از یک سیستم نرم افزاری ،طیفها را تجزیه و تحلیل میکند و توالی مولکول DNA را نشان میدهد.

توالی یابی به روش ماکسام وگیلبرت
همزمان با ابداع روش توالی یابی سنگر، روش توالی یابی ماکسام و گیلبرت که به روش توالی یابی شیمیایی نیز شناخته شده بود، وارد دنیای ژنتیک شد . در ابتدا روش ماکسام و گیلبرت از استقبال بهتری نسبت به روش سنگر برخوردار شدند، زیرا در روش ماکسام و گیلبرت، محققان میتوانستند از DNA تخلیص شده به صورت مستقیم استفاده کنند، در حالی که اساس شروع به کار متد سنگر کلونینگ ژن است. اما با گذر زمان متد ماکسام- گیلبرت با کاهش استقبال رو به رو شد.
روش ماکسام-گیلبرت شامل چهار مرحله است:
- در ابتدا مولکول DNA تک رشته را دناتوره میکنند و بعد در انتهای ’۵ آن با استفاده از فسفر 32 برچسب گذاری انجام میدهند.
- سپس DNA توسط پیپریدین و دو ترکیب شیمیایی دیگر که به پورینها و پیریمیدینهای مولکول DNA حمله میکنند، شکسته میشود. این ترکیبات شیمیایی منجر به برش مولکول DNA در قسمت یکی از ۴ باز آدنین، تیمین، گوانین و سیتوزین میشود که با قرار دادن چهار لوله آزمایش برای هر ترکیب شیمیایی، میتوان ۴ نمونه از قطعات با برشهای یکسان را به دست آورد.
- در مرحله بعدی هر کدام از آن بر روی ژل پلی آکریلامید با درصد بالا مورد الکتروفورز قرار میگیرند که در این قسمت برای تمایز بین قطعات مختلف از برچسبهای رادیواکتیو استفاده میشود.
- برای خوانش توالی DNA نمونه، باید از کوچکترین قطعه در پایین ژل شروع کرد.
نسل دوم توالی یابی
به تدریج روشهای دیگری جهت ارتقاء سطح سرعت و افزایش بازدهی تعیین توالی DNA اختراع شده است که Next Generation Sequencing نامیده شد که در آن ها نیازی به ژل و کلونینگ نیست و از واکنش زنجیرهای پلیمراز جهت افزایش قطعات استفاده میکنند که نتیجه آن سرعت و بازدهی بالا میباشد .
روش توالی یابی Pyrosequencing
با توسعه علوم در حیطه ی نانوتکنولوژی و بیوانفورماتیک روشهای ثابت دیگری جهت افزایش سرعت و افزایش بازدهی تعیین توالی DNA اختراع شده است. در همین دوره با پیشرفت توالی یابی دی دئوکسی در مقیاس بزرگ، متد دیگری که تفاوت چشمگیری با متدهای قبلی داشت پدید آمد.
اختلاف چشمگیر این متد با روشهای موجود به دلیل dentsهای لیبل دار رادیواکتیو یا فلئورسنت بود. در اصل دانشمندان از روش لومینسانس برای اندازگیری سنتز پیروفسفات استفاده کردند. این روش شامل پروسه ی دو آنزیمی بود: در واکنش اول ATP سولفوریلاز پیروفسفات را به ATP تبدیل و سپس ATP تولید شده همچون سوبسترا برای آنزیم لوسیفراز مصرف و لوسیفراز تولید نور هماهنگ با مقدار پیروفسفات میکند. این نشان های نوری بوسیله دوربینهای حساس به سیگنالهای ساطع شده موسوم به CCD بهصورت یک پیک ثبت میشود. نوکلئوتیدهایی که در هر گام اضافه میشوند مشخص و برابر هستند، توالی DNA را در حین سنتز را قادر خواهید بود بر اساس تولید این پیک، بدست آورید. آنزیمی به نام آپیراز که یک نوع آنزیم نوکلئوتیداز میباشد ، در هر گام نوکلئوتیدهای قبلی را برای اضافه کردن نوکلئوتیدهای جدید از محیط حذف میکند.
بدیهی است با وجود تفاوتها در دو روش سنگر و pyrosequencing اساس توالی یابی در هر دو روش سنتز رشته اسید نوکلئیک است که تکینیکهای SBS نامیده میشوند. زیرا هر دو نیاز به عمل مستقیم DNA پلیمراز برای تولید و مشاهده محصول دارند که این متد برخلاف روش ماکسام و گیلبرت است .
در مطالب زیر به بررسی روش pyrosequencing که توسط Pal Noreen و همکارانش پی ریزی شد میپردازیم:
- استفاده از دئوکسی نوکلئوتیدهای طبیعی
- از الکتروفورز که پروسه ای طولانی و وقت گیر است استفاده نمیشود و نتایح در همان لحظه قابل مشاهده است.
- این متد نیاز به کلونینگ را مرتفع کرده و از پروسه PCR جهت ازدیاد قطعه مورد نظر استفاده میکند که نتیجه ی آن افزایش سرعت و بازدهی تعیین توالی DNA است.
روش pyro sequencing در سال 2005 توسط شرکت 454 Life Sciences و بعدها توسط شرکت Roche خریداری شد و به اولین تکنولوژی تجاری تحت عنوان NGS تبدیل شد. از آن پس اصطلاح NGS جهت توصیف مجموعهای از فناوریها با قدرت بالاتر در مقایسه به توالییابی به شیوه سنگر مورد استفاده قرار گرفت .
روش توالی یابی ( Semiconductor Sequencing (Ion Torrent
در این روش که از یک سیستم شناسایی نیمه رسانا و همچنین از emPCR برای تکثیر قطعات استفاده میشود، در سال 2010 توسط شرکت Life Technologies مطرح شد. مراحل آمادهسازی و تعیین توالی این تکنیک به pyrosequencing بسیار شبیه میباشد.در روش Ion Torrent تعیین توالی برخلاف روش نوری که در سایر سیستمها استفاده میشود، از شناسایی یون هیدروژن که در هنگام سنتز DNA آزاد میشود، استفاده میکند. اگر نوکلئوتیدی که به محیط وارد میشود، مکمل نوکلئوتیدهای DNA هدف باشد، یک یون هیدروژن آزاد میشود. در نتیجه یک واکنش در حسگر فوق حساس یونی رخ میدهد.این روش با حذف نیاز به مواد فلورسنت و دوربین سبب افزایش سرعت توالییابی و کاهش هزینهها شده است. بعدها دومین دستگاه تعیین توالی این شرکت با نام Ion proton را در سال 2012 عرضه شد که میزان خروجی آن افزایش چشمگیری داشت .
روش توالی یابی ایلومینا ( Illumina (Solexa
این تکنیک توسط شرکت ایلومینا در سال 2007 ارائه گردید. دراین فن آوری از ملکولهای آداپتوری استفاده شد که روی یک سطح جامد ثابت شده بودند. روند کار به این صورت است که در ابتدا ژنوم مورد نظر به قطعات DNA صد تا صد و پنجاه جفت بازی شکسته میشود. سپس با اتصال به دو انتهای هر قطعه آداپتور قطعات، تک رشتهای میشوند.قطعات روی سطح جامدی که با توالیهای مکمل آداپتور پوشیده شده است، پراکنده میشوند و در انتها هر قطعه از DNA بر روی سطح جامد پایدار باقی میماند.در مرحله بعد با اتصال انتهای آزاد این قطعات نیز به توالیهای مکمل آداپتور یک بریج تشکیل میدهند. سپس تکثیر این بریج با افزودن مواد مورد نیاز PCR آغاز میشود که این فرآیند، bridge amplification نام گرفته است. پس پایان هر سیکل، DNA دو رشتهای حاصل دناتوره شده تا دوباره PCR انجام گیرد. این فرایند تا زمانی که تعداد زیادی از قطعه مورد نظر ایجاد شود، تکرار میشود. پس از اتمام مرحله تکثیر، محصولات PCR باید دناتوره شوند.
برای مطالعه بیشتر توالی یابی نسل سوم بخوانید



