ارتباط مستقیم با کارشناس فروش
توالی یابی DNA | روش های توالی یابی از گذشته تا امروز

توالییابی DNA یکی از پیشرفتهترین فناوریهای زیستمولکولی است که به دانشمندان امکان میدهد تا ساختار ژنتیکی موجودات زنده را با دقت بالا بررسی کنند. با تعیین دقیق ترتیب نوکلئوتیدهای DNA، میتوان اطلاعات ژنتیکی را تحلیل کرده و به فهم بهتر بیماریها، تکامل گونهها و ساختار ژنها دست یافت. پیشرفتهای سریع در تکنولوژی توالییابی، از روشهای کلاسیک مانند سنگر تا نسلهای جدید و سوم، انقلابی در علوم زیستی و پزشکی ایجاد کرده است که زمینه ساز تحقیقات نوین و درمانهای هدفمند شدهاند.
توالییابی DNA (تعیین سکانس)
توالییابی DNA (DNA Sequencing) فرایندی است که ترتیب نوکلئوتیدهای آدنین (A)، گوانین (G)، سیتوزین (C) و تیمین (T) در مولکول DNA مشخص میشود. DNA اطلاعات ژنتیکی موجودات زنده را حمل میکند و تعیین توالی آن، امکان بررسی ارتباطات ژنتیکی، روابط فیلوژنتیکی و جهشهای ژنتیکی را فراهم میکند.
اهمیت تعیین توالی DNA
براساس نظریه فرانسیس کریک، توالی نوکلئوتیدهای DNA مستقیماً بر توالی اسیدهای آمینه و در نتیجه پروتئینها تاثیر دارد. توالییابی ژنومها باعث درک بهتر ساختار و عملکرد سلولها و ارگانیسمها شده است. در گذشته تعیین توالی کامل ژنوم انسانی چندین سال و میلیاردها دلار هزینه داشت؛ اما امروزه این فرایند با هزینهای کمتر از هزار دلار و در چند ساعت انجام میشود.
روشهای توالییابی DNA
1. روش سنگر (Sanger Sequencing)
این روش مبتنی بر استفاده از نوکلئوتیدهای دیدئوکسی (ddNTP) است که باعث خاتمه زنجیره DNA در محلهای خاص میشوند. DNA هدف با یک آغازگر (پرایمر) به همراه DNA پلیمراز و مخلوطی از نوکلئوتیدهای طبیعی و ddNTP در واکنش قرار میگیرد. زمانی که ddNTP وارد رشته میشود، سنتز متوقف شده و قطعات DNA با طولهای متفاوت ساخته میشوند.
قطعات سپس توسط الکتروفورز ژل یا کپیلاری الکتروفورز جدا میشوند و با توجه به طول قطعات و برچسب فلورسنت، توالی DNA مشخص میگردد. این روش دقیق است ولی برای توالییابیهای بزرگ کند و پرهزینه است.
شکل زیر ، یک واکنش توالی با ddATP الکتروفورز میشود. هر خط یک مولکول DNA از یک طول خاص را نشان میدهد که در نتیجه یک واکنش پلیمریزاسیون است که با اضافه کردن یک نوکلئوتید ddATP خاتمه یافته است. ستونهای دوم، سوم و چهارم شامل ddCTP ، ddTTP، ddGTP بودند.
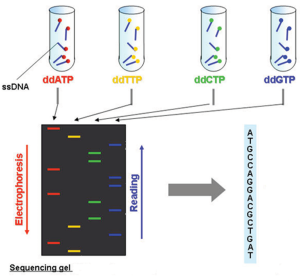
توالی یابی به روش سنگر
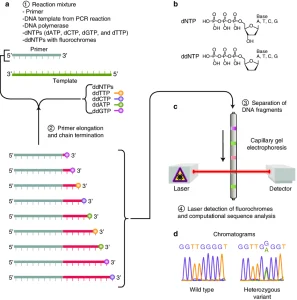
با گذشت زمان، تغییرات این روش با وجود یک برچسب فلورسنت متفاوت در هر ddNTP بود. در واقع برای آغازگر دیگر منبع برچسب رادیواکتیو یا فلورسنت استفاده نشد. بنا براین متد سنگر همان روش توالی یابی رنگ پایان دهنده میباشد، که از چهار رنگ با طیف انتشاری بدون همپوشانی برای هر یک از ddNTP استفاده میکند.
همانطور که در تصویر مشاهده میکنید رنگ خاتمه دهنده از یک طرح کلی توالی یابی را مشاهده میکنید، یک مخلوط واکنش منفرد با تمام فاکتورهای مورد نیاز برای گسترده سازی مولکول DNA را دارد. مخلوط واکنش همچنین حاوی غلظتهای کمی از چهار ddNTPs با برچسب فلورسنت متفاوتی هستند. پس از اتمام واکنش توالی یابی، محصول را بر روی ژل کپیلاری الکتروفورز میبرند و با آنالیز طیف انتشار از هر باند DNA روی ژل نتایج این فرایند بدست میآید. سپس با استفاده از یک سیستم نرم افزاری ،طیفها را تجزیه و تحلیل میکند و توالی مولکول DNA را نشان میدهد.

2. روش ماکسام-گیلبرت (Maxam-Gilbert Sequencing)
این روش شیمیایی DNA را در بازهای آدنین، تیمین، گوانین و سیتوزین به صورت انتخابی برش میدهد. قطعات حاصل بر اساس اندازه جدا شده و با برچسبهای رادیواکتیو یا فلورسنت توالی مشخص میشود. به دلیل استفاده از مواد شیمیایی سمی و پیچیدگی روش، امروزه کمتر کاربرد دارد.

نسل دوم توالییابی (Next Generation Sequencing – NGS)
این فناوریها توانستند با توالییابی همزمان میلیونها قطعه DNA، سرعت و بازدهی را افزایش دهند و هزینهها را کاهش دهند.
روشهای مهم NGS:
Pyrosequencing: با اندازهگیری نور تولید شده در واکنش آنزیمی که پیروفسفات آزاد میکند، توالی DNA مشخص میشود. نیاز به الکتروفورز ندارد و سریع است.
Ion Torrent: به جای روش نوری، تغییرات پH ناشی از آزاد شدن یون هیدروژن هنگام افزودن نوکلئوتیدها را اندازهگیری میکند. حذف نیاز به فلورسنت و دوربین، سرعت و هزینه را کاهش داده است.
Illumina: بر پایه تکثیر DNA روی سطح جامد (bridge amplification) و خواندن فلورسانس هر نوکلئوتید در هر چرخه است. این روش بسیار دقیق و پرکاربردترین فناوری NGS است.
نسل سوم توالییابی (Third Generation Sequencing)
این نسل با تمرکز بر خواندن مستقیم مولکولهای بلند DNA، بدون نیاز به تکثیر PCR، سرعت و دقت را افزایش داده است.
فناوریهای نسل سوم:
PacBio SMRT: خواندن زمان واقعی توالی DNA با استفاده از آنزیم DNA پلیمراز، مناسب برای شناسایی جهشهای پیچیده و ساختار ژنوم.
Oxford Nanopore: عبور DNA از نانوپور و اندازهگیری تغییرات جریان الکتریکی برای تعیین توالی. این فناوری قابل حمل است و توالیهای بلند را به راحتی میخواند.
اهمیت و کاربرد توالییابی DNA
توالییابی DNA پایه بسیاری از پیشرفتهای زیستشناسی و پزشکی است. فناوریهای نوین به تشخیص سریع بیماریهای ژنتیکی، تحقیقات دقیقتر و درمانهای شخصیسازی شده کمک میکنند. همچنین این فناوریها امکان مطالعه دقیقتر ارتباطات ژنتیکی و تکامل گونهها را فراهم کردهاند.
برای مطالعه بیشتر توالی یابی نسل سوم بخوانید




